最近在《自然通讯》上发表的一项研究中,一个团队提出了一种基于人工智能的方法,以探讨蛋白质结构的相似性和关系。该团队成员来自弗吉尼亚大学,包括数据科学学院院长菲尔·伯恩(Phil Bourne)、资深科学家卡姆·穆拉(Cam Mura)以及最近的校友伊莱·德莱岑(Eli Draizen)。
他们的研究颠覆了传统的蛋白质结构关系观念,揭示了许多传统方法未能捕捉到的微弱关系。
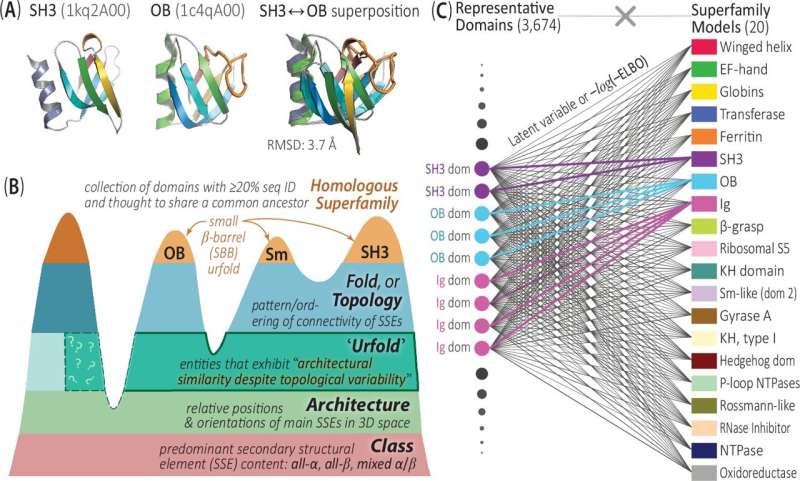
具体而言,作者介绍了一个计算框架,能够在大规模(涵盖无数蛋白质)下检测和量化这些关系,结合了深度学习方法与一种新的概念模型(称为Urfold),使得不同拓扑结构的蛋白质也能展现出结构相似性。
Bourne、Mura和Draizen与Stella Veretnik共同完成了这一项目。所有作者均为伯恩和穆拉计算生物科学实验室的成员,该实验室隶属于数据科学学院和弗吉尼亚大学生物医学工程系。
该研究成果是伯恩实验室多年来开发的名为DeepUrfold的人工智能框架的结晶,使得Urfold结构关系理论得以系统性和大规模的探索。
通过DeepUrfold,伯恩实验室的研究团队在蛋白质宇宙中发现了许多微弱的结构关系,这些关系在进化或其他方面被认为是无关的。
在捕捉和描述这些遥远关系时,DeepUrfold从“社区”的视角审视蛋白质关系,避免了将蛋白质划分为孤立、不重叠的类别的传统方法。总体而言,这些新方法推动研究者超越静态、几何的蛋白质相似性思考,走向更为综合的研究方式。
伯恩是数据科学学院的创始院长,以其在结构生物信息学和计算生物学领域的研究而闻名。在职业生涯早期,他共同领导了RCSB蛋白质数据库的开发,这一数据库为蛋白质结构信息的获取提供了重要支持,并为AlphaFold等现代人工智能的发展奠定了基础。
Mura在UVA数据科学学院和生物医学工程系任职,拥有结构和计算生物学的广泛背景,包括基于RNA的系统的生化和晶体学研究以及DNA的分子生物物理学。他从分子进化的角度研究生物系统,并探索这些领域与数据科学的交集。
Draizen在伯恩的指导下获得弗吉尼亚大学生物医学工程博士学位,目前在加州大学旧金山分校担任计算生物学博士后研究员。Veretnik是弗吉尼亚大学的高级研究科学家,专注于计算生物学及蛋白质折叠的结构、功能和进化。
本文来自作者[公仙仙]投稿,不代表5664玩立场,如若转载,请注明出处:https://5664wan.cn/keji/202412-6052.html


评论列表(4条)
我是5664玩的签约作者“公仙仙”!
希望本篇文章《人工智能驱动的方法挑战了传统的蛋白质结构观点》能对你有所帮助!
本站[5664玩]内容主要涵盖:国足,欧洲杯,世界杯,篮球,欧冠,亚冠,英超,足球,综合体育
本文概览: 最近在《自然通讯》上发表的一项研究中,一个团队提出了一种基于人工智能的方法,以探讨蛋白质结构的相似性和关系。该团队成员来自弗吉尼亚大学,包括数据科学学院院长菲尔·伯恩(Ph...